
近日,来自斯坦福大学的研究团队在《Nature Biotechnology》上发表了一篇题为“Inferring gene expression from cell-free DNA fragmentation profiles”的文章,基于cfDNA测序的多基因表达水平分析建立了一种分析cfDNA片段化模式的新方法,即“Epigenetic Expression Inference from Cell-free DNA-sequencing (EPIC-seq)”技术,可同时应用于癌症早期检测、分型和伴随诊断等方面。

研究背景
细胞游离DNA(cfDNA)是释放到血浆中的降解的DNA片段,存在于人体的各种体液中,随组织损伤、癌症和炎症反应等发生浓度变化。正常人体的cfDNA主要通过细胞凋亡过程中产生的小而均匀的185~200 bp小片段DNA。而肿瘤坏死的组织细胞由于非正常的凋亡过程,导致产生大小不同且大于200 bp的大片段DNA。1947年,Mandel和Metais首次报道了外周血中存在cfDNA。生理情况下,血液中的cfDNA主要来源于白细胞的坏死和凋亡。在某些疾病和特殊状态下,其他细胞也会释放游离DNA入血,包括来源于肿瘤细胞的循环肿瘤DNA(ctDNA)和来源于胎儿的cfDNA等。
血液中循环肿瘤DNA (ctDNA)检测为非侵入性癌症检测带来希望。染色质的片段化特征曾被用来推断细胞游离DNA (cfDNA)的基因表达谱,但目前这种片段化方法需要高浓度的肿瘤源性DNA,且分辨率有限。
研究方法
研究人员将启动子片段描述为一种表观基因组cfDNA特征,它可以预测单个基因的RNA表达水平,通过对目标基因启动子进行靶向测序可以推断出表观遗传表达水平。通过对201例癌症患者和87例健康成年人的共329份血液样本的分析,展示了肺癌和弥漫性大B细胞淋巴瘤的亚型分型水平的研究。另外,研究人员应用EPIC-seq技术对接受PD-(L)1免疫治疗的患者的多时间点连续采样血液样本进行研究,发现其推断的基因表达谱与患者临床表现存在密切关联。这些工作表明,EPIC-seq技术可以实现无创、高通量的肿瘤组织起源分析,在诊断、预后和治疗领域具有巨大潜力。
研究结果
cfDNA分子特征与基因表达水平的关联
研究人员假设,受活性启动子调控的cfDNA片段与非活性启动子调控相比,存在更多的随机切割模式的可能性,从而可实现从cfDNA推断单个基因表达,反映了包括实体瘤在内的不同组织来源的各种细胞类型的分型。通过对未知来源的肿瘤患者样本的深度全基因组测序分析,发现了定位到高表达基因的cfDNA分子与低表达基因相比显示出了更多的片段长度多样性特征。研究人员进一步比对了肺癌患者和健康对照样本,确定了启动子片段熵与非小细胞肺癌(NSCLC)基因表达水平显著相关。在NSCLC中高表达的基因在肿瘤患者血液中的启动子片段熵明显升高,并且与肺癌分期有关,而低表达基因则观察不到这种趋势,表明这种cfDNA特征具有基因和组织特异性。
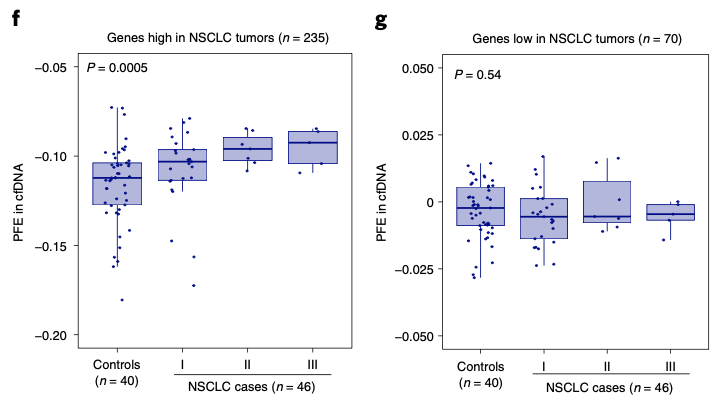
实体肿瘤中验证cfDNA推断基因表达水平
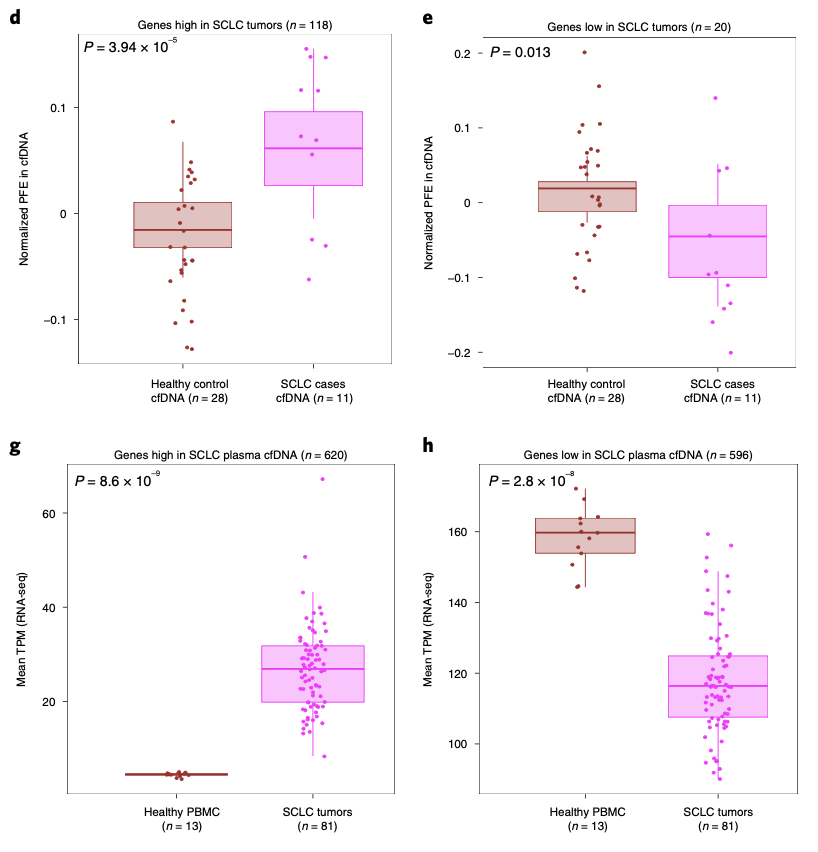
研究人员进一步验证全外显子测序是否可以进一步验证cfDNA推断表达水平的准确度。通过对11例小细胞肺癌(SCLC)患者和28例健康对照者血浆cfDNA进行超深度全外显子测序,作者发现对于原发SCLC肿瘤中高表达的肿瘤基因,肿瘤患者的血浆cfDNA启动子片段熵特征明显高于健康对照组,而在SCLC肿瘤中低表达的基因则表现出相反趋势。研究人员将这两种特征表达基因合并计算得到一个可以有效区分SCLC癌症患者和健康对照组的计算模型(AUC=0.98)。在这些特征表达基因中,研究人员进一步筛选确定了三种SCLC特征标志物基因ASCL1,ANK1和ASTN1。
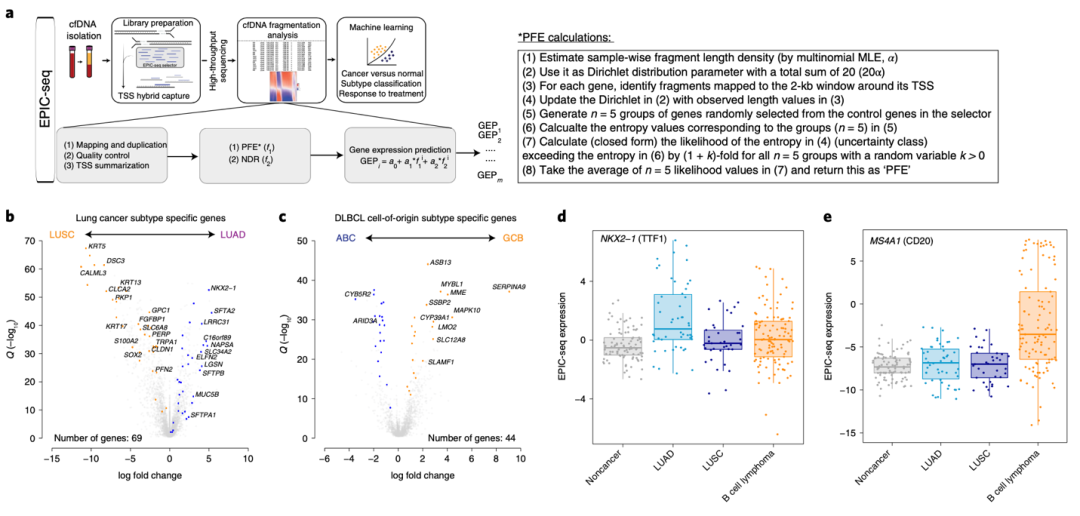
cfDNA片段化谱推断基因表达水平(EPIC-seq技术)
测序深度的增加提高了cfDNA推断基因表达水平的准确性,因此作者开发了一种基于cfDNA的TSS侧翼区域的定向深度测序与机器学习相结合的EPIC-seq技术,可以精准预测基因表达水平。为了验证这一概念,研究人员分别将EPIC-seq技术应用于实体肿瘤的非侵入式组织学分型和血液肿瘤的分子分型中。首先,鉴定出肿瘤组织中高表达且在全血样本中相对低表达的基因,然后通过分别对比肺腺癌(LUAD)和肺鳞状细胞癌(LUSC),GCB型和ABC型弥漫性大B细胞淋巴瘤(DLBCL)中的差异表达,从而减低当初肿瘤亚型特异性的基因。研究人员利用EPIC-seq流程共分析了来自288例成年人的373个血浆样本cfDNA,其中包括来自87例NSCLC患者的109个血浆样本,114例弥漫性大B细胞淋巴瘤的126个血浆样本和来自87例健康对照者的94个血浆样本。最终鉴定出LUAD中高表达的特征基因NKX2-1可有效实现肺癌组织分型,在DLBCL中高表达的MS4A1(CD20)基因可有效实现淋巴瘤的免疫表型分型。
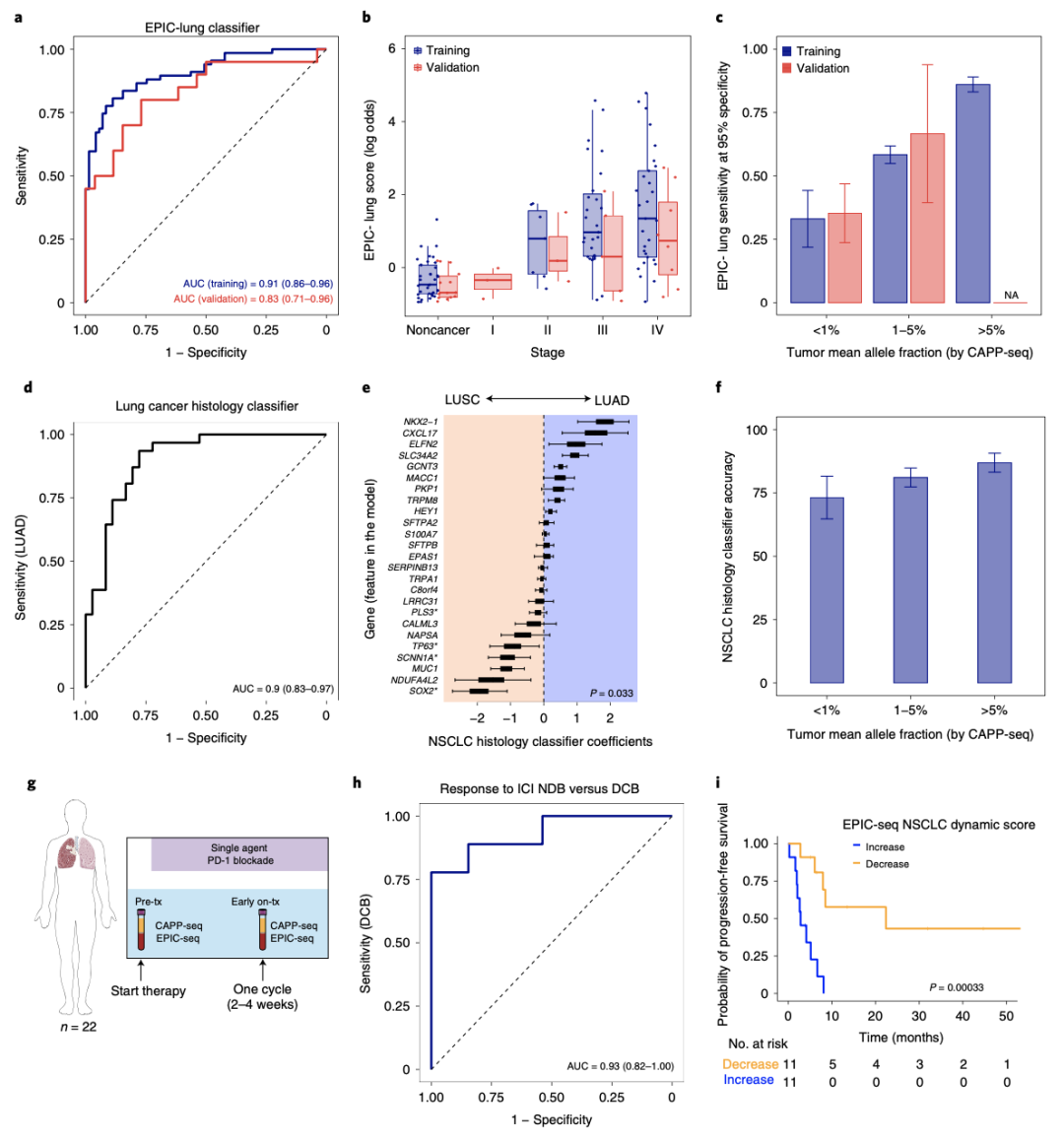
EPIC-seq用于肺癌的非侵入式检测、分型与PD-(L)1免疫治疗疗效预测
研究人员通过机器学习方法对肺癌患者与健康对照样本进行模型分析,将队列随机分成训练集和验证集。其中训练集包含67例肺癌患者(7例II期,30例III期,30例IV期)和71例健康对照,验证集包含20例肺癌患者和23例健康对照,训练集和验证集中的AUC分别为0.91和0.83,展现出了良好的肺癌检测效果,且III- IV期中晚期肺癌患者的EPIC分数与II期相比有所升高。为了进一步研究EPIC-seq方法在肺癌分型方面的应用,研究人员选择了一个67例肺癌患者对例,根据组织分型分为肺腺癌组(36例)和肺鳞癌组(31例),并利用训练和交叉验证方法得到了良好的分型效果(AUC=0.90)。对于晚期NSCLC患者,PD-(L)1免疫治疗十分重要,然而其药效与毒副作用的研究仍不明确,目前缺乏有效可靠的生物标志物用于在治疗前或治疗早期精准预测患者能否从免疫治疗中获益。因此,研究人员用EPIC-seq方法分析了22例接受PD-(L)1免疫治疗的NSCLC患者治疗前和治疗开始前4周的血液样本,开发了可有效预测治疗效果的“肺动态因数”。
EPIC-seq用于弥漫性大B细胞淋巴瘤(DLBCL)的非侵入式检测和分子分型
DLBCL是一种常见的非霍奇金淋巴瘤,具有显著的临床和生物学异质性。这种异质性在临床上可通过国际预后指数(IPI)、原发肿瘤活检的基因分型等方法获得信息,但仍然缺乏有效的低侵入式检测方法。研究人员将EPIC-seq技术应用于分析91例DLBCL患者和71例健康对照的血液cfDNA,训练出一种表现良好的区分模型(AUC=0.92),在验证集(23例DLBCL和23例健康对照)中AUC达到0.96,且EPIC分析与IPI具有显著的分级相关性。进一步分析了DLBCL患者接受治疗前后的数据,证实了EPIC-seq方法可以通过分侵入式手段检测出组织来源的DLBCL信号,并真实反映出治疗前后的临床表现。DLBCL根据细胞来源分成两种不同的分型,其中GCB型患者通常具有更好的预后效果并有可能预测其靶向治疗的敏感性。因此,研究人员对治疗前的样本进行进一步的EPIC-seq分析,得出了能够良好区分出GCB型的模型分数,与基于肿瘤组织水平的分型结果具有良好的相关性(r=0.84)。
总结与展望
研究人员介绍了一种利用cfDNA片段模式对基因表达进行无创推断的方法即“EPIC-seq”技术,可广泛应用于临床相关应用,包括肿瘤检测、亚型分类、疗效评估和预后分析等。为了最大限度地利用这种片段化技术,仍有必要进行进一步的细化研究。局部肿瘤患者存在相当大的分子异质性,低ctDNA水平可能会阻碍其直接用于早期癌症检测。随着使用这种方法研究更多的基因和更多的片段组特征,可以得到更强的信号进行小肿瘤沉积的非侵入性检测,从而实现更多特定的信号通路活动和组织病理研究。
参考文献:
Mohammad Shahrokh Esfahani, Emily G. Hamilton et al. Inferring gene expression from cell-free DNA fragmentation profiles. Nature Biotechnology, 2022, 40: 585-597.