

蛋白质组学作为一种研究蛋白质在生物体内功能和调控的手段,已获得广泛应用。结合人工智能技术,特别是机器学习方法,能够提供更加准确、有效的蛋白质分析及预测方法,为癌症治疗带来新的突破。本文将探讨人工智能助力蛋白质组学在癌症中的应用,包括蛋白质组学数据分析、蛋白质(肽)与 蛋白质相互作用以及个性化治疗等方面,并通过实例和结果展示其在癌症研究中的潜力。
01 蛋白质组学数据分析
蛋白质组学研究的基础在于对蛋白质组学数据进行分析,这对于挖掘相关信息具有关键性意义。在蛋白质组学数据分析中,人工智能技术的应用涵盖了数据整合、特征提取以及模型构建等多个方面。例如,Gonçalves, Emanuel et al.(2022)使用数据非依赖采集(DIA)-质谱法(MS)方法,基于深度学习的深蛋白组标记(DEEBAR)算法对949个人类细胞系的泛癌蛋白组图进行分析,并成功识别出1538个新的生物学标志物[1]。Zhang, Fangfei et al.(2020)提出了一种创新的表型预测技术,利用深度神经网络对质谱原始数据进行分析。以 102 个肝癌样本的良性和恶性分类为例,测试数据的准确性可达 96.8%[2]。研究结果显示,机器学习有助于在复杂的蛋白质组学数据中发现重要的生物信息。
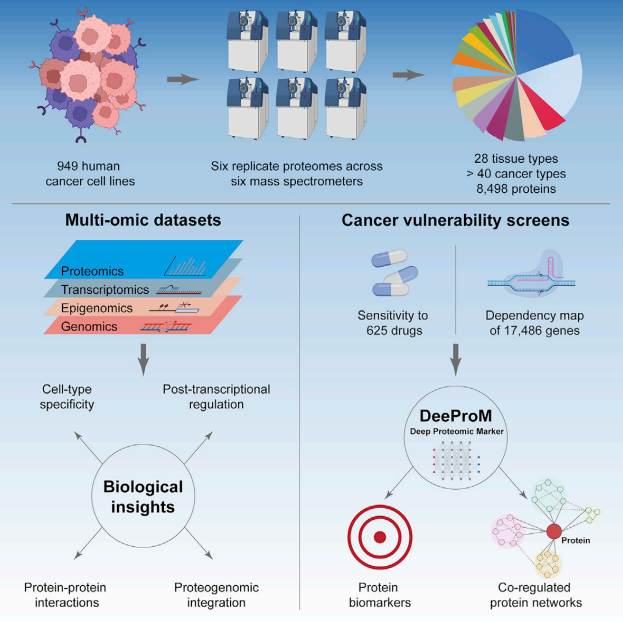
图1 基于深蛋白组标记(DEEBAR)算法的
泛癌蛋白组分析流程图
02 蛋白质与蛋白质相互作用的分析
癌症是一种高度复杂的疾病,涉及多种信号通路,其中癌蛋白扮演着重要角色并介导大量致癌蛋白-蛋白相互作用(PPIS)。得益于人工智能和机器学习的应用,可以从大规模蛋白质组学数据中构建和分析PPIS网络。例如:Kim, Minkyu et al.(2021)采用不同的机器学习评分算法,构建了一个与乳腺癌相关的蛋白质相互作用网络,从而发现了一些新的潜在靶点[3]。
03 肽与蛋白的相互作用预测
肽与蛋白质之间的相互作用对于各种基本细胞功能具有重要意义,识别这些相互作用,对于发现与癌症相关的多肽药物至关重要。目前,主要有两种主流方法用于预测肽与蛋白质的相互作用,即基于序列方法及基于结构方法。这些方法通常需要借助机器学习技术进行特征提取和模型构建。例如,CGKronRLS[4] 软件采用基于核的回归算法,而 Liu, Yong et al.开发的 NRLMF 算法[5] 以及清华大学曾坚阳教授和赵诞提出的深度学习框架 (CAMP)[6]等。
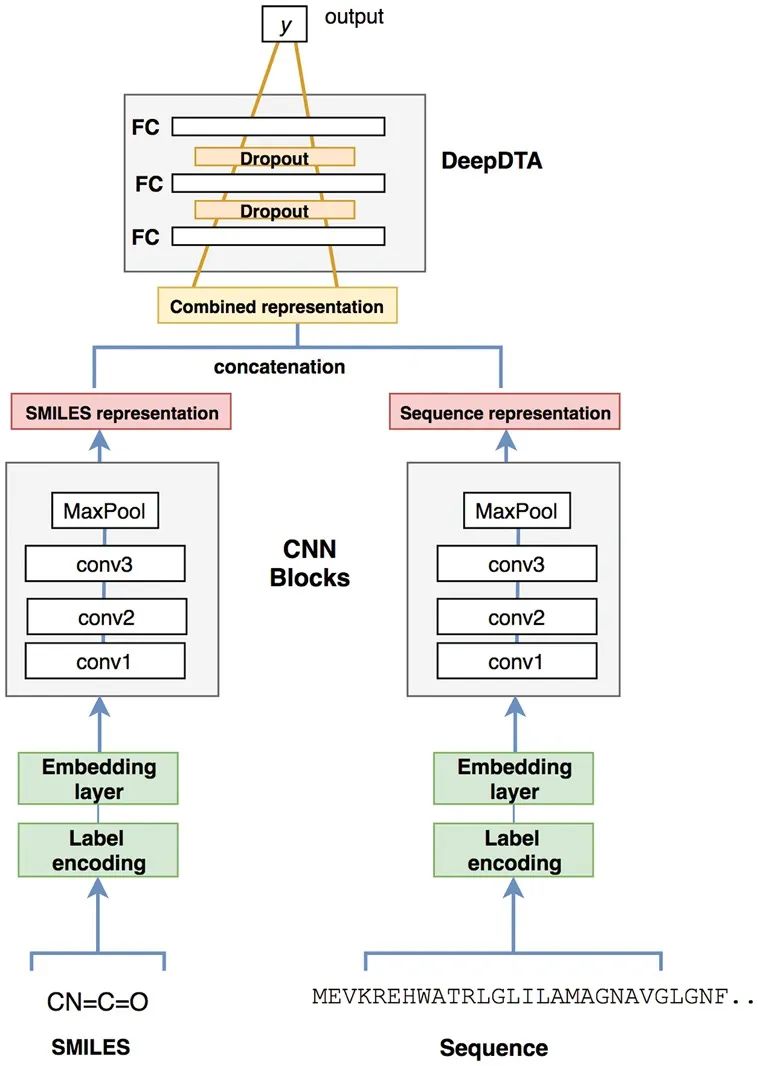
图2 DeepDTA模型流程图
04 个性化治疗
癌症的个性化治疗是一种根据患者的个体差异制定的治疗策略。蛋白质组学数据分析和机器学习在个性化治疗中发挥着重要作用。例如,Ferrarotto, Renata et al.(2021)利用反相蛋白阵列技术(Revers Phase Protein microArray,RPPA)对唾液腺样囊性癌样本进行 K-均值聚类,建立分子亚型,并为每种分子亚型确定治疗靶点,实现了个性化治疗目标 [7]。
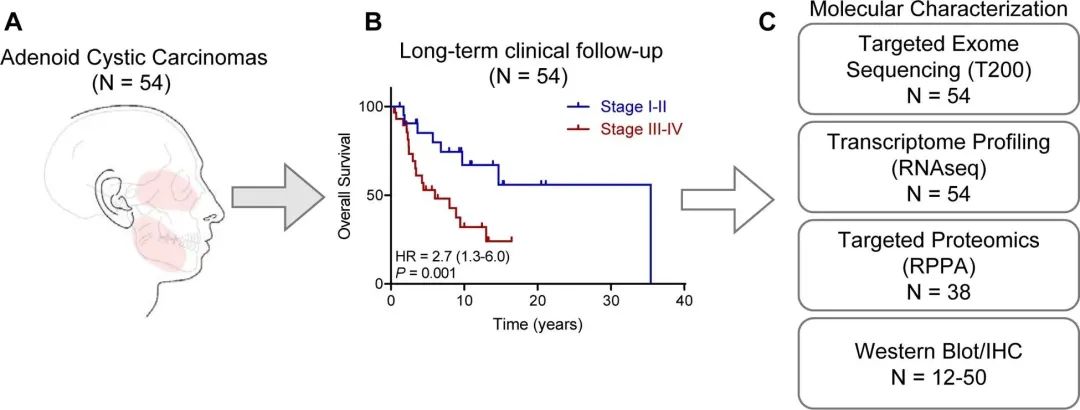
图3 唾液腺样囊性癌样本分子亚型分析流程
在这些研究中,机器学习扮演了至关重要的角色。通过对大量蛋白质组学数据的分析和挖掘,机器学习算法能够识别出与癌症相关的蛋白质特征,预测蛋白质的相互作用关系,并帮助确定个体化治疗方案,其高效和准确性大大提高了癌症治疗的成功率。
然而,机器学习方法在蛋白质组学应用中仍面临一些挑战。首先,需要大规模的、高质量的蛋白质数据来训练和验证模型。其次,在模型设计和参数选择方面仍需进一步优化。此外,机器学习结果的解释和验证也是一个重要的问题。
参考文献:
1. Gonçalves, Emanuel et al. “Pan-cancer proteomic map of 949 human cell lines.” Cancer cell vol. 40,8 (2022): 835-849.e8. doi:10.1016/j.ccell.2022.06.010.
2. Zhang, Fangfei et al. “Phenotype Classification using Proteome Data in a Data-Independent Acquisition Tensor Format.” Journal of the American Society for Mass Spectrometry vol. 31,11 (2020): 2296-2304. doi:10.1021/jasms.0c00254.
3. Kim, Minkyu et al. “A protein interaction landscape of breast cancer.” Science (New York, N.Y.) vol. 374,6563 (2021): eabf3066. doi:10.1126/science.abf3066.
4. Cichonska, A. et al. Computational-experimental approach to drug-target interaction mapping: a case study on kinase inhibitors. PLoS Comput. Biol. 13, e1005678 (2017).
5. Liu, Y., Wu, M., Miao, C., Zhao, P. & Li, X.-L. Neighborhood regularized logistic matrix factorization for drug-target interaction prediction. PLoS Comput. Biol. 12, e1004760 (2016).
6. Lei, Yipin et al. “A deep-learning framework for multi-level peptide-protein interaction prediction.” Nature communications vol. 12,1 5465. 15 Sep. 2021, doi:10.1038/s41467-021-25772-4.
7. Ferrarotto, Renata et al. “Proteogenomic Analysis of Salivary Adenoid Cystic Carcinomas Defines Molecular Subtypes and Identifies Therapeutic Targets.” Clinical cancer research : an official journal of the American Association for Cancer Research vol. 27,3 (2021): 852-864. doi:10.1158/1078-0432.CCR-20-1192.